今年度の学位記授与式がありました。新領域創成科学研究科(先端生命科学専攻)から中坪さんと黄さんが修士の学位を授与されました。コロナ禍の人数制限で安田講堂に入れなかったので、3人でお祝いのランチに行きました。



東京大学大気海洋研究所 微生物グループ Microbial Oceanography lab, aori utokyo
スプーン一杯の海水から探る地球環境 Explore the microbial world in a drop of seawater
今年度の学位記授与式がありました。新領域創成科学研究科(先端生命科学専攻)から中坪さんと黄さんが修士の学位を授与されました。コロナ禍の人数制限で安田講堂に入れなかったので、3人でお祝いのランチに行きました。


キャンパスの隣の柏の葉公園にて、研究室メンバーの送別のランチ会をしました。桜はまだ一分咲きといったところでしたが、気持ちの良い天気で和やかに送別の歓談をすることができました。



大幅改造で生まれ変わった「白鳳丸」の慣熟航海に乗船してきました
我が国を代表する研究船「白鳳丸」ですが、建造から30年を経過して老朽化が指摘されてました。新たな白鳳丸の建造はなかなか難しかったのですが、関係者の努力により、エンジン4台の交換やウインチの増設などの大改造が実現しました。これであと20年は活躍してくれそうです。エンジンを全交換したおかげで、スピードと静粛性がアップして、以前にも増して使い勝手が良さそうです。航走速度がアップすると、観測点までの到達時間が短くなり、その分長く滞在して観測することができますので、研究者にとっては重要です。見た目や内装はほとんど変わっていませんが、煙突に排気処理装置が付いたために四角い形となりました。鹿児島を出港して、東シナ海を南下しながら屋久島沖、沖縄本島沖で観測して、太平洋側に抜けてから琉球海溝で観測して再び鹿児島に戻りました。2/20から3/3までおよそ2週間の航海でした。






今年も波の花サンプリングに行ってきました。
前週までの大雪が嘘のような晴れ続きで、しかも到着の翌日にタイミングよく波の花が取れるという例年になく順調な滑り出しでした。それ以降も波の花が発生して2回のサンプリングができました。今回は、高見研究員のアイデアで、波の花の酵素活性測定やそれらの生産菌の分離も行いました。今後の研究の新たな展開が期待できる良い材料を得ることができました。帰る日から再び雪が降り始めましたので、今回はなんともタイミングの良い調査となりました。
 今年の波の花
今年の波の花
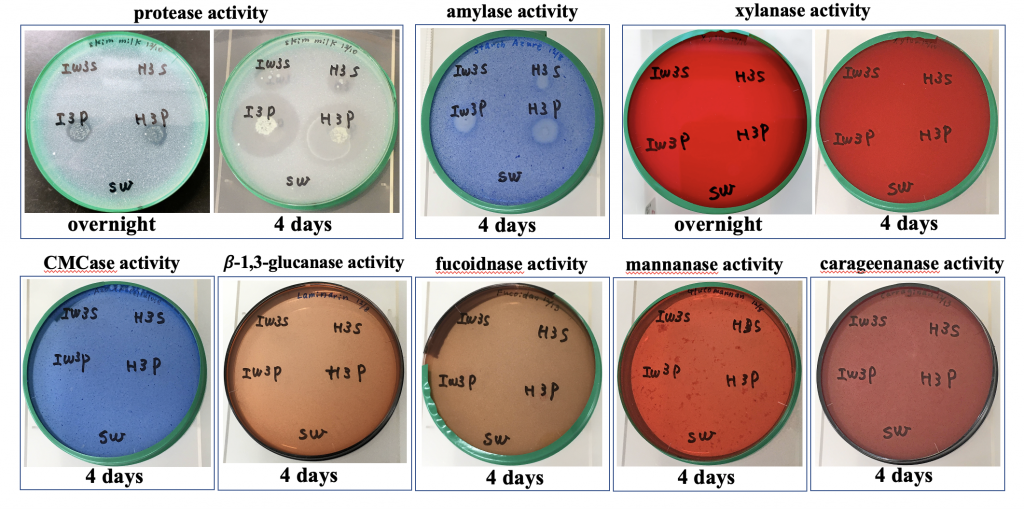 酵素活性検出のためのプレートアッセイ(写真:高見英人博士)
酵素活性検出のためのプレートアッセイ(写真:高見英人博士)
今年も深海海洋保護区の調査のために、「かいめい」に乗船して小笠原諸島の西側に位置する西七島海嶺周辺海域に行ってきました。10/12に横須賀のJAMSTEC岸壁を出港して10/25に戻ってきました。昨年は小笠原で下船しましたが、今回はそうした寄港はなく残念でした。調査の方はとても順調で、良い航海でしたが、すでに全国的に陽性者が激減していた時期だったにも関わらず、コロナ対策として乗船1週間前からホテルで隔離生活を余儀なくされたのには閉口しました。昨年とは違う海山でサンプリングできましたので、さらにデータを加えて解析できそうです。ちなみに、昨年12月に初めて指定された沖合海底域いわゆる深海底の海洋保護区は4箇所、伊豆・小笠原海溝、中マリアナ海嶺・西マリアナ海嶺北部、西七島海嶺、マリアナ海溝北部となっています。

 メタゲノム解析用の堆積物サンプリング
メタゲノム解析用の堆積物サンプリング
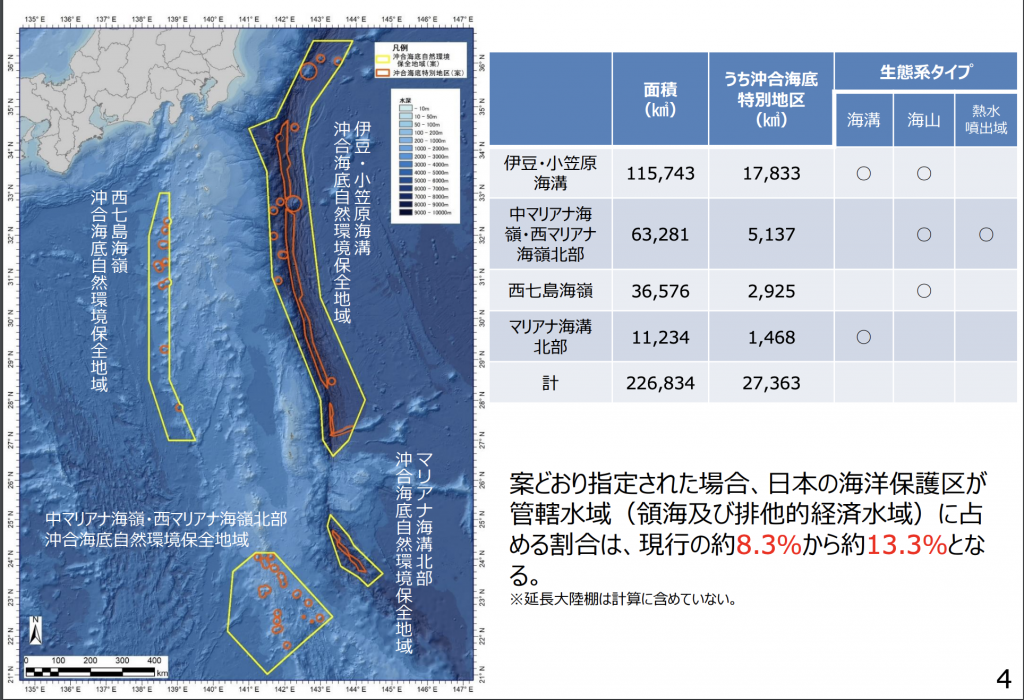
環境省資料より
9月24日(金)本郷キャンパスの鉄門記念講堂にて、ダイキン社と東大の協創活動の一環であるUGIPの成果発表会が開催されました。2年前の夏に視察に行った海外インターンシッププログラムですが、残念ながらコロナ禍のため2年連続で海外に行けず国内での実施となりました。昨年は単発のワークショップのみでしたが、今年は海外駐在社員とのオンラインミーティングなどを交えながら1ヶ月間かけてガッツリとダイキン社から提示された課題に取り組みました。サイエンスと同じく新しい価値を作るという点で、ビジネスの世界ではどうアプローチするのか興味深く勉強させていただきました。実生活との関わりの中でかつ国際情勢を踏まえながら多様な海外市場に対してどうアプローチするのかを考えるのは、サイエンスとは違った面白さがあるなあと感じました。来年は海外でのインターンシップが復活することを願います。余談ですが、高市早苗さんの自民党総裁選挙出馬会見やご著書の中で、実学重視の人材育成活動の好事例として、このプログラムを挙げられていました。日本の大学生の海外志向の弱さが指摘されている中で、同様の取り組みが広く展開されることを期待していますし、今後も微力ながら東京大学の活動をお手伝いできればと思います。



8月24日~26日の3日間、インターンシップ「新種の微生物を見つけよう!遺伝子で紐解く微生物の種多様性」を実施しました。特任研究員の高部さんと院生の長谷川さんが全体のマネージメントをやってくれて、春と同じ内容で、ただし違うサンプルの単離株の16SrRNA遺伝子配列を決定して、分類群の特定を行いました。今回は学部1年から4年生まで色々な大学から6名の学生さんが参加してくれました。使った単離株は、波の花、真鶴の海水、潮溜まりの海水、水月湖の海水から分離したものをそれぞれ一株ずつ選んでもらい、各自4株の遺伝子配列をサンガー法で決定しました。デモ用に解析した株も含めて、全部で33株の配列を決定し、そのうち新種判別の基準である既知種との相同性97%以下の株は5株でした。そのうち3株は真鶴沖の海水から分離したもの、2株は能登半島の曽々木海岸で採集した波の花から分離したものでした。今後、チャンスがあれば新種記載することになるかもしれません。


8月最初の週に広島大に行ってきました。1ヶ月ほど前に来年度の研究費申請のための計画をいろいろ思案しているうちに、簡単な予備実験を思いつきました。私たちは、微生物活動がエアロゾルや雲核生成過程にどう影響するかをテーマに研究を行っています。例えば、微生物分解によって有機物の化学形態が変わりますが、そうした変化は有機物を含むエアロゾルの雲核生成能(CCN活性)に影響するのか?というのが基本的な疑問です。まずは、基質とその分解産物をそれぞれエアロゾル化してCCN活性を測定すれば一つの答えが出ます。ということで、どうしても確かめておきたくなり、エアロゾル発生装置と雲凝結核活性測定装置のある共同研究者のところに押しかけたというわけです。心配していたトラブルもなく、なんだか予想以上にそれらしいデータが取れて大満足でした。最終日には大崎下島のMitarai Baseで今後の打ち合わせをして帰ってきました。御手洗地区は、江戸時代に、風待ち潮待ちの港町として栄えた町ということで、商家、茶屋、船宿など当時の風情が今でも残されています。お米屋さんの店舗兼住宅だったというMitarai Baseもそうした町並みの中にある素敵な建物で、海を望む部屋で心地よい潮風を受けながらの贅沢なミーティングでした。

広島大学

エアロゾル発生装置とCCNカウンター

MitaraiBase
高部由季特任研究員が、大槌湾や宇和海で分離した好気性光合成細菌のドラフトゲノムを、米国微生物学会が発行するMicrobiology Resource Announcement誌で公開しました。
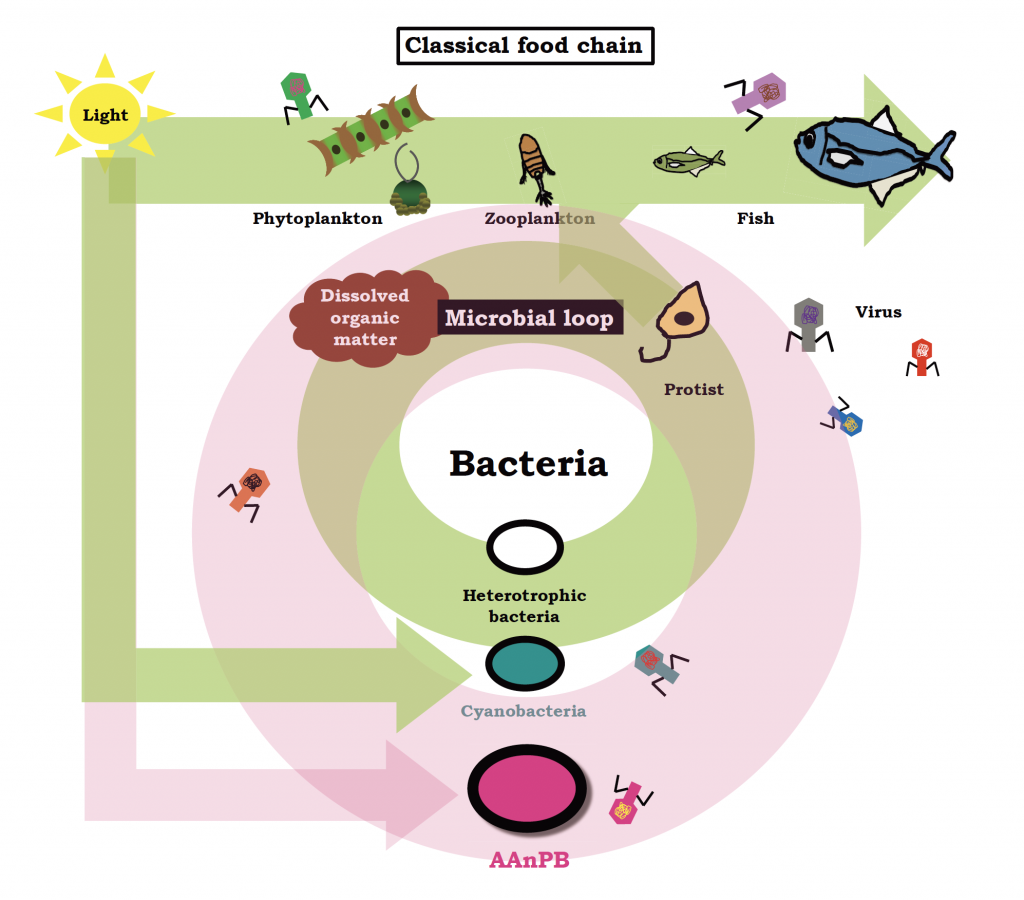
酸素非発生型好気性光合成細菌 (Aerobic Anoxygenic Phototrophic Bacteria, AAnPB) は、海洋表層に普遍的に分布し、増殖速度が速いため、微生物ループを介した炭素循環におけるキープレイヤーとして重要です。今回ゲノムを解読したのは、岩手県大槌湾の海水から分離培養したRoseobacter sp. OBYS 0001株と、愛媛県愛南町の真鯛養殖いけす周辺の海域から分離したJannaschia spp. AI_61とAI_62株です。
Roseobacter sp. OBYS 0001株は、16S rRNA遺伝子の配列相同性(100%)からR. litoralisと考えられます。この株は、高部博士のこれまでの研究(Sato-Takabe et al., 2012; 2014)で、光合成に関する生理的性状が詳しく調べられています。今回のゲノムデータは、それらの性状がどのような遺伝的機能によって維持されているのかや、他の光合成細菌との進化系統学的な関係がどうなっているのかなどを明らかにする研究に利用されることになります。
一方、愛南町の養殖いけす周辺海域には、上記AAnPBが通年で分布し、時に全菌数の24%超を占めることがわかっています(Sato-Takabe et al., 2016; 高部, 2020)。Jannaschia spp. AI_61とAI_62株のゲノムデータは、魚類養殖場の低次生態系を構成する主要メンバーの遺伝的機能を明らかにする意味で重要です。また、Jannaschia属として現在記載されている12種のうち、光合成能が確認されているのは2種しかありません。今回のAI_61とAI_62株は、最近縁種との16S rRNA遺伝子配列相同性(96.53%)から、本属の新種の可能性もあり、新たな光合成能をもつJannaschia属の株として、比較ゲノム解析をすると面白そうです。
好気性光合成細菌についてさらに詳しく知りたい方は、高部博士の総説をぜひご一読ください

6月30日に渋谷キューズで開催したイベント「OceanDNAテック2021」が、おかげさまで盛況のうちに終了しました。当日はオンラインと会場でのハイブリッド開催でしたが、時節柄か渋谷駅直結という便利な会場立地にも関わらず圧倒的にオンライン参加の多いイベントとなりました。事前登録は300名超で、実際の参加は270名ほどでした。この手のワークショップとしては、まずまずの集客ではなかったかと思います。大学関係は3割ほどで、民間企業や一般の方に多く参加いただいたようですので、自動化技術の社会実装を目指すワークショップとしては良いアピールができました。
さらに広くアピールするために、録画した動画を公開しました。こうした動画を簡単に公開できるのは、オンラインワークショップならではですね。社会実装に向けて、引き続き情報発信を続けていきます。

