高部由季特任研究員が、大槌湾や宇和海で分離した好気性光合成細菌のドラフトゲノムを、米国微生物学会が発行するMicrobiology Resource Announcement誌で公開しました。
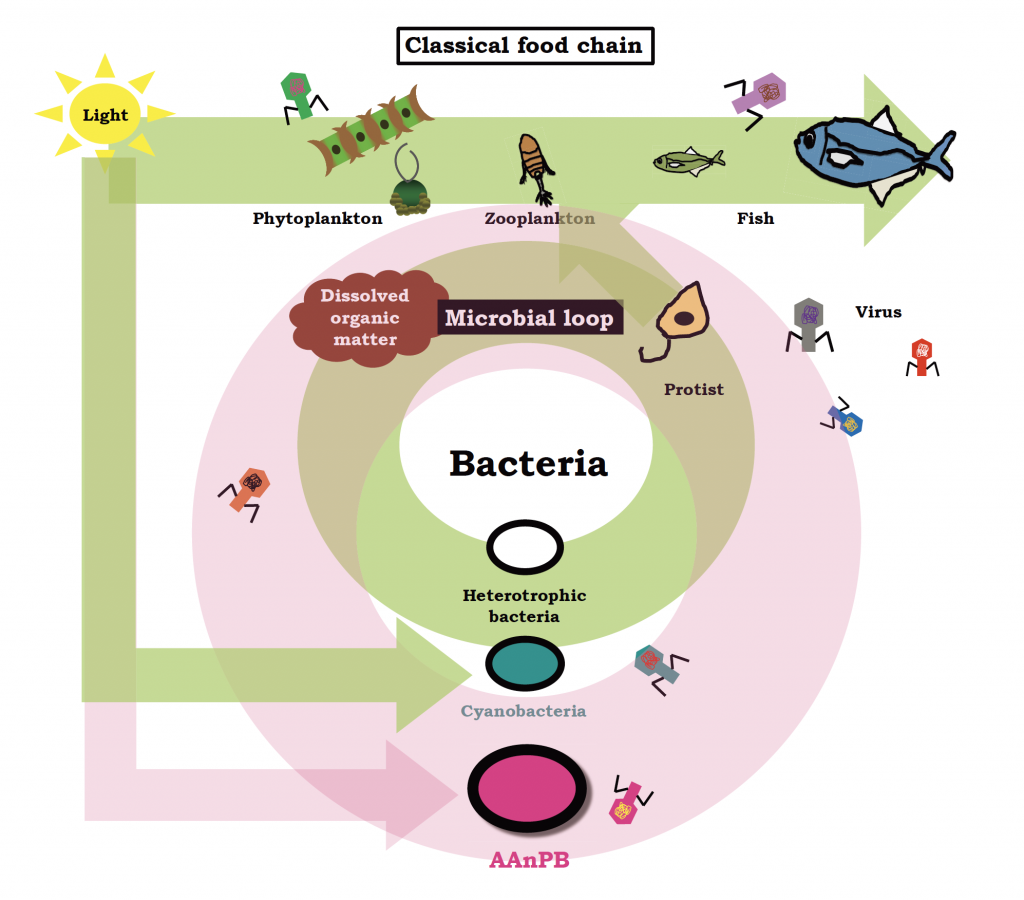
酸素非発生型好気性光合成細菌 (Aerobic Anoxygenic Phototrophic Bacteria, AAnPB) は、海洋表層に普遍的に分布し、増殖速度が速いため、微生物ループを介した炭素循環におけるキープレイヤーとして重要です。今回ゲノムを解読したのは、岩手県大槌湾の海水から分離培養したRoseobacter sp. OBYS 0001株と、愛媛県愛南町の真鯛養殖いけす周辺の海域から分離したJannaschia spp. AI_61とAI_62株です。
Roseobacter sp. OBYS 0001株は、16S rRNA遺伝子の配列相同性(100%)からR. litoralisと考えられます。この株は、高部博士のこれまでの研究(Sato-Takabe et al., 2012; 2014)で、光合成に関する生理的性状が詳しく調べられています。今回のゲノムデータは、それらの性状がどのような遺伝的機能によって維持されているのかや、他の光合成細菌との進化系統学的な関係がどうなっているのかなどを明らかにする研究に利用されることになります。
一方、愛南町の養殖いけす周辺海域には、上記AAnPBが通年で分布し、時に全菌数の24%超を占めることがわかっています(Sato-Takabe et al., 2016; 高部, 2020)。Jannaschia spp. AI_61とAI_62株のゲノムデータは、魚類養殖場の低次生態系を構成する主要メンバーの遺伝的機能を明らかにする意味で重要です。また、Jannaschia属として現在記載されている12種のうち、光合成能が確認されているのは2種しかありません。今回のAI_61とAI_62株は、最近縁種との16S rRNA遺伝子配列相同性(96.53%)から、本属の新種の可能性もあり、新たな光合成能をもつJannaschia属の株として、比較ゲノム解析をすると面白そうです。
好気性光合成細菌についてさらに詳しく知りたい方は、高部博士の総説をぜひご一読ください


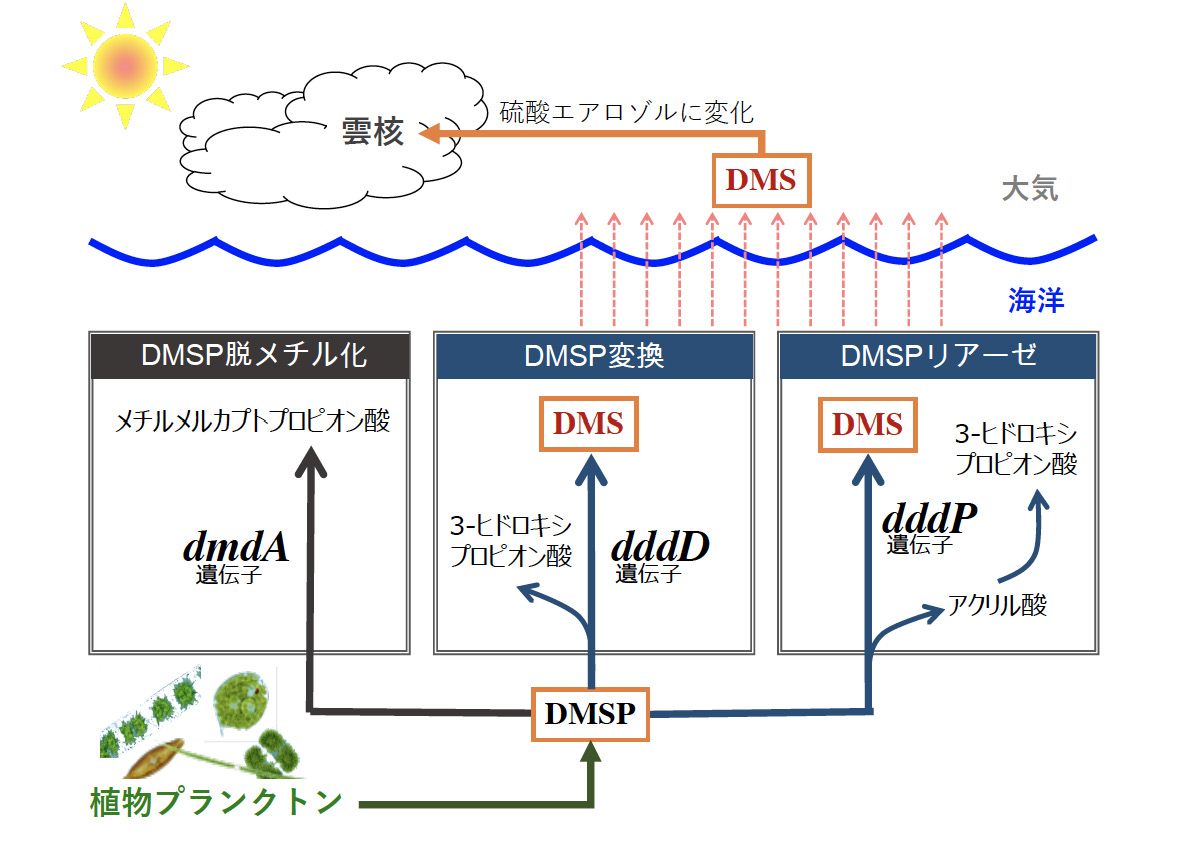 バクテリアによるDMSP代謝経路と雲生成への影響
バクテリアによるDMSP代謝経路と雲生成への影響 メソコズム実験:200Lタンク4基を屋外水槽に入れて温度を一定に保つ(右)数日後に植物プランクトンが大増殖し緑色に変化したタンク内の海水(左)
メソコズム実験:200Lタンク4基を屋外水槽に入れて温度を一定に保つ(右)数日後に植物プランクトンが大増殖し緑色に変化したタンク内の海水(左)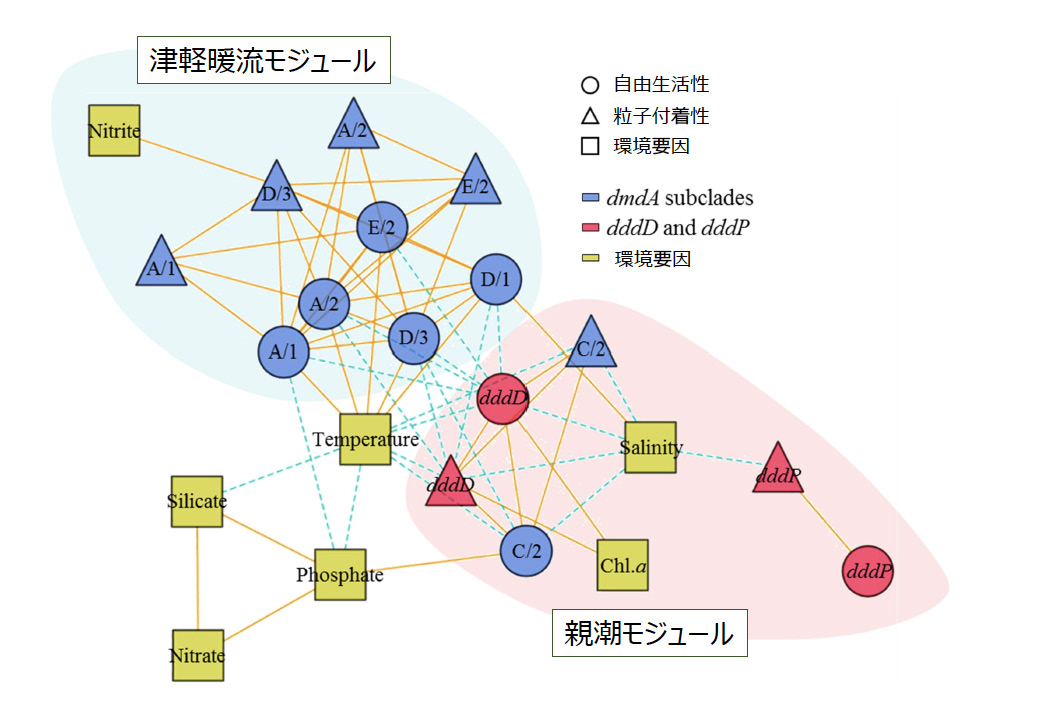 DMSP代謝関連遺伝子と環境要因の相関ネットワーク図:機能遺伝子単位でのまとまりが見られる。実線は正相関、破線は負相関を示す。水温と正相関を示すdmdA遺伝子群は津軽暖流の影響を強く受けており、水温や塩分と負相関でクロロフィル濃度と正相関を示すdddDとdddP遺伝子は親潮の影響を受けていると考えられる
DMSP代謝関連遺伝子と環境要因の相関ネットワーク図:機能遺伝子単位でのまとまりが見られる。実線は正相関、破線は負相関を示す。水温と正相関を示すdmdA遺伝子群は津軽暖流の影響を強く受けており、水温や塩分と負相関でクロロフィル濃度と正相関を示すdddDとdddP遺伝子は親潮の影響を受けていると考えられる